Выполнены исследования по анализу полиморфизма микросателлитных локусов в генетических ресурсах плодовых культур Юга России. На основе полученной экспериментальной информации создана пополняемая база данных SSR-фингерпринтов изученных генотипов, включающая информацию об идентифицированных аллелях, использованных в работе SSR-маркеров.
Ключевые слова: генетическое разнообразие, коллекции генетических ресурсов, Malus, Prunus, Pyrus, микросателлитные ДНК-маркеры, мультиплексный анализ.
The analysis of polymorphism of microsatellite loci in the genetic resources of fruit crops in southern Russia have been done. On the base of experimental data the newly created database SSR-fingerprints of studied genotypes, including information about identified alleles used in SSR-markers was established
Keywords:genetic diversity, collections of genetic resources, Malus, Prunus, Pyrus, microsatellites, multiplex analysis.
Изучение биологического разнообразия является одним из наиболее важных научных направлений в генетике культурных растений. В настоящее время наиболее эффективными методами для изучения генетического разнообразия, выяснения филогенетических взаимосвязей на различных таксономических уровнях являются методы, основанные на использовании ДНК-маркеров. К наиболее информативным ДНК-маркерам относят микросателлитные последовательности ДНК (SSR). К одному из главных направлений наших исследований, наряду с изучение генетического разнообразия плодовых культур является формирование генетической базы данных ДНК-паспортов образцов из коллекций генофонда плодовых культур Северного Кавказа, включающей SSR-фингерпринты изученных генотипов.
Материалом для исследований являются современные сорта плодовых культур и автохтонные сорта Северного Кавказа, и других регионов РФ, а также республик бывшего СССР, представленные в коллекциях генетических ресурсов Юга России. Кроме того, в исследования вовлечены видовые образцы указанных родов плодовых культур. Исследования выполняются при научной кооперации с МОСВИР (г. Майкоп) и Дагестанской опытно селекционной станцией (г. Буйнакск). В работе используется 25 SSR маркеров для рода Malus; 22 для рода Prunus; 19 для рода Pyrus. Фрагментный анализ выполняется на автоматическом генетическом анализаторе ABI prism 3130, с последующей обработкой данных в программе Gene Mapper 4.1.
Результаты исследований.
В ходе выполнения исследований SSR-маркеры, используемые в работе, с учетом температуры отжига праймеров и диапазона размеров продуктов амплификации были сгруппированы в мультиплексные наборы, представленные в таблице 1.
Для каждого маркера в мультиплексном наборе были использованы специфичные флуоресцентные красители: FAM, R6G, ROX или TAMRA, имеющие разные оптические спектры флуоресценции, что позволяет проводить в одной реакции одновременную идентификацию нескольких фрагментов. В таблице 1 представлены данные о мультиплексных наборах, сформированных и апробированных в ходе выполнения исследований.
Таблица 1
SSR-маркеры и мультиплексные наборы, используемые для SSR-паспортизации генофонда плодовых культур
|
Malus |
Prunus |
Pyrus |
|
1)CH01d03+CH02c02a+SdSSR 2)CH05e03+CH03a04+CN581493 3)Hi16d02+CH04e03+CN445290 4)СH01h10+СH01h01+CH01f03b+ СH02c06 5)VfC+CH05f06+CH04e05+CH02d08 6) Hi02C07+GD147+CH01f02+CH02c11 7) CH04c07+GD12+CH03d07+CH02C09 |
1) CPSCT004+BPPCT007+BPPCT028 2) UDP98-412+BPPCT037+BPPCT023 3) CPPCT044+CPPCT040 4) BPCCT002+Ps12a02a+UDP98-409 5) CPPCT022+UDP98-407+UDP98-410 6) UDP98-022+EMPaS06+UDP96-018 7) UDP96-008+BPPCT038+CPPCT006 8) EMPaS02+EMPaS01 |
1) EMPc108+EMPc117+EMPc115 2) GD142+BGT23b 3) СH01h01+CH01f07a+Ch01d08 4) CH03d12+EMPc11+CH03g07 5) CH05C06+CH02B10+Nh011b 6) NH044a+NH015a+GD96
|
Мультиплексный анализ позволяет выполнять анализ одновременно по нескольким локусам (от двух до четырех), что значительно повышает экономическую эффективность SSR-генотипирования. На рисунке 1, для примера приведен пример фрагментного анализа на автоматическом генетическом анализаторе ABIprism 3130 по одному из мультиплексных наборов.
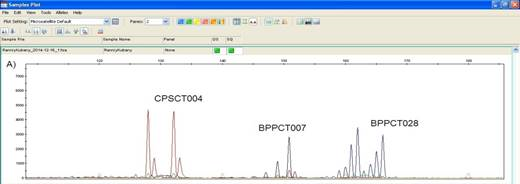
Рис. 1. Результаты фрагментного анализа сорта персика Ранний Кубани по мультиплексному набору, включающему SSR — маркеры CPSCT004, BPPCT007 и BPPCT028
Два пика/фрагмента по маркерам CPSCT004 и BPPCT028 свидетельствует о гетерозиготности микросателлитных локусов, присутствие только одного фрагмента по маркеру BPPCT007 — о гомозиготности.
Для выполнения SSR-генотипирования создан пополняемый банк образцов ДНК. На данный момент он включает порядка 530 образцов ДНК сортов и сортовых образцов плодовых: из них 190 сортов и 17 видовых образцов рода Malus, 98 сортовых и 16 видовых образцов рода Pyrus, около 200 сортовых образцов рода Prunus.
Результаты SSR-генотипирования, после обработки первичных данных фрагментного анализа с использованием программы Gene Mapper 4.1 вносятся в базу данных. Для каждого образца представлена информация о дате отбора и экстракции ДНК из журнала регистрации образцов (столбцы А, B), наименование образца (название сорта, видового образца) — столбец D и размеры аллелей SSR-локусов в парах нуклеотидов (Рисунок 2).
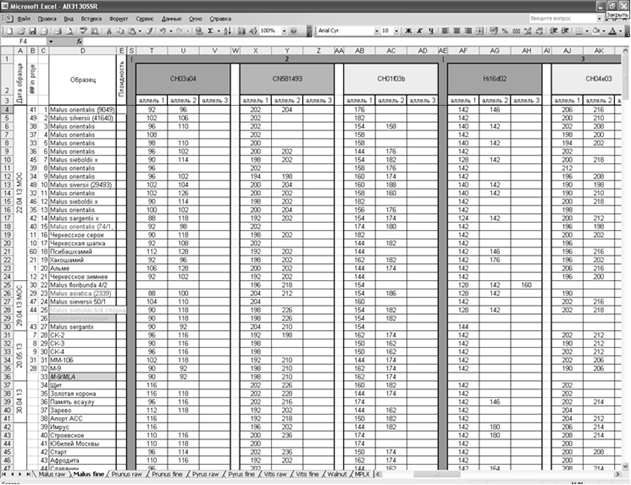
Рис. 2. Принтскрин страницы базы данных накопления и анализа результатов SSR- генотипирования
Наличие базы данных SSR-фингерпринтов позволяет в дальнейшем эффективно решать вопросы, связанные с генетической идентификацией образцов при возникновении спорных вопросов о генетической идентичности образцов, сортовой принадлежности посадочного материала. Кроме того, полученные данные используются в настоящее время для анализа генетического разнообразия коллекций генетических ресурсов изучаемых видов плодовых культур.







